VT tenía la historia desde el principio. Ahora que la CIA tiene oficialmente la tarea de encontrar una fuente de COVID, alguien a quien culpar, el vasto programa estadounidense de defensa de la guerra biológica que crea todos los virus desagradables que hemos visto, probablemente el ébola, ciertamente la gripe porcina, y luego los libera “accidentalmente”. de tal manera que se cumpla la agenda de la CIA.
La riqueza está centralizada.
El mundo está aterrorizado.
Actualización:� VT ha estado rastreando las estadísticas de COVID y la desinformación / censura desde el primer día. Ahora, a la luz del ataque de Beirut (Trump nos respalda), ahora vemos el lanzamiento de una versión híbrida de COVID 19 en el área metropolitana de Nueva York como un ataque terrorista.
Las estadísticas sobre hospitalizaciones y muertes allí han sido modificadas / censuradas, lo que demuestra que se trataba de un ataque biológico. Las tasas de mortalidad fueron muchas veces más altas que en otros lugares e incluso se dirigieron específicamente a la población judía, algo que Israel ha hecho antes.
Hemos observado la manipulación de Internet de Google Corporation, los hemos rastreado y encontramos evidencia clara y absoluta que vincula a los que ejecutaron el ataque de Beirut con los que lanzaron COVID 19 en los EE. UU. Y el mundo.
También hemos visto videos de Beirut censurados por Facebook, Google / YouTube y Twitter y coordinamos con MSM para vender una narrativa falsa que involucra fertilizantes inexplorables mientras se acumula evidencia de un ataque masivo no solo por parte de Israel, sino también de un brillante conjunto de fintas, drones, aviones, e incluso un misil.
Quién sabe, una bomba nuclear podría haberse cargado allí con facilidad desde un camión y nadie se daría cuenta.
Ahora, con Beirut en ruinas, los Emiratos Árabes Unidos uniéndose al Gran Israel y la monstruosidad de Qanon vendiendo un estado policial a la chusma, lo inexplicable se vuelve claro.
A continuación, demostramos la creación de COVID, un proyecto de la CIA realizado a través de un programa de cobertura en una universidad privada utilizando expertos en armas biológicas. Ahora lo vemos liberado, ¿el objetivo? Saquear la economía estadounidense, llevar a Estados Unidos a una guerra civil y permitir que el poder se centralice bajo el control de Kosher Nostra sin el subterfugio, ahora innecesario.
Este artículo contiene pruebas sólidas que no se pueden cuestionar ni negar, que puede presentar a cualquier agencia gubernamental o profesional de la salud.
Lo que aún no se ha probado, pero se está enfocando es que el programa de armas biológicas de Estados Unidos en Fort Detrick, Maryland, el equipo y ciertamente el personal clave, ciertamente migraron a laboratorios secretos en las grandes universidades estatales para “esconderse a la vista”.
Siga las carreras, se incluyen todos los enlaces, de quienes trabajaron en el proyecto Wuhan-COVID en 2017.
Además, tenga en cuenta que exactamente el mismo personal y equipo se utiliza para la investigación y prueba de “prevención” falsa como armamento y producción real.
Desde que se escribió este artículo, hemos comenzado a analizar las operaciones mundiales del contratista nuclear / bio / químico de EE. UU., Battelle, el favorito de Kushner-Trump, y sus laboratorios secretos en todo el mundo.
Cuando comenzamos, nuestra gente empezó a recibir amenazas. Eso fue un grave error.
Envíe este documento a cualquier médico u otro especialista calificado en biociencias. Mira lo que dicen.
Introducción
Los documentos a continuación mostrarán que la investigación para crear COVID 19 comenzó en los Estados Unidos en 2006 y culminó en un arma biológica exitosa en 2015, con trabajo realizado en la Universidad de Carolina del Norte y en Harvard y en el laboratorio de la Administración de Alimentos y Medicamentos en Arkansas. .
Su trabajo se tituló:
Un grupo de coronavirus de murciélago similar al SARS muestra potencial para la emergencia humana
Hicieron esto y más, tanto más como leerás a continuación.
Como dijo Trump, una y otra vez, los chinos estuvieron involucrados.
El Laboratorio clave de patógenos especiales y bioseguridad, el Instituto de Virología de Wuhan, la Academia de Ciencias de China, Wuhan, China, suministró el virus del murciélago de Wuhan que se utilizó en el estudio estadounidense. Su nombre fue incluido solo por esa razón.
COVID 19 fue un proyecto de armas biológicas del ejército de los EE. UU. Para fabricar una enfermedad que causa neumonía y que sería casi imposible de vacunar en pacientes mayores de 40 años.
La prueba está aquí, simplemente desplácese hacia abajo. El estudio fue realizado por la Universidad de Carolina del Norte y financiado por USAID / CIA. Eligió un virus de murciélago chino y decidió incluir también un centro médico en Wuhan.
Ahora sabemos por qué, una cortina de humo de la culpa de un programa con el que China tuvo poco o nada que ver, algo satánicamente malvado y puramente estadounidense.
En noviembre de 2015, se publicó un estudio que describe la capacidad de producir el virus con el que estamos tratando ahora. Entre los muchos involucrados se encontraba un laboratorio en Wuhan, China. Fue catalogado desde el principio como una de las docenas, en su mayoría estadounidenses, que trabajan en este proyecto.
Sin embargo, un participante clave quedó fuera, USAID. Se sospecha, profundamente, que USAID es un frente para la investigación estadounidense de guerra biológica como la realizada en Tbilisi, Georgia y otros lugares, muy documentada. Esta es la cita que agrega a USAID al grupo de financiamiento de la investigación.
20 de noviembre de 2015
En la versión de este artículo publicada inicialmente en línea, los autores omitieron reconocer una fuente de financiamiento, financiamiento de USAID-EPT-PREDICT de EcoHealth Alliance, a Z.-LS El error se ha corregido para las versiones impresas, PDF y HTML de este artículo. .
[Nota del editor:�ahora presentaremos el artículo sesgado de Pravda y, más abajo, el estudio real que demuestra la capacidad de producir COVID 19, lo que demuestra que no es un virus natural de una vez por todas.
En cuanto a quién hizo qué, este no es nuestro trabajo, pero estamos demostrando, categóricamente, que cuando se menciona un laboratorio chino, es un jugador menor en un esfuerzo estadounidense, como se describe exhaustivamente a continuación.
Esto hace que discutir sobre el laboratorio de Wuhan sea posiblemente cómplice de la guerra biológica.
De manera similar, cuando la revista Forbes y otros afirmaron que podían probar que COVID 19 se hizo de forma natural y, por supuesto, tenían el mismo acceso que nosotros, sospechamos que son parte de un esfuerzo de desinformación vinculado a USAID y la guerra biológica.
La sospecha no es prueba. La prueba es una prueba y no hay prueba suficiente para ahogar. Nuestras gracias a los profesionales médicos estadounidenses que PIMPED a sí mismos al Ejército de Estados Unidos y la CIA y que nos ayudó a donde estamos ahora, una nación rota en pedazos …�VeteransToday]
Pravda.Ru: Dicho material apareció en 2015 en el sitio web de la revista científica Natura en 2015. Luego, los autores afirmaron que después del advenimiento del virus del SARS (2002-2003) y el síndrome respiratorio de Oriente Medio (MERS), los científicos estaban conscientes del riesgo de transmisión interespecífica que daría lugar a una epidemia entre las personas.
De manera similar, cuando la revista Forbes y otros afirmaron que podían probar que COVID 19 se hizo de forma natural y, por supuesto, tenían el mismo acceso que nosotros, sospechamos que son parte de un esfuerzo de desinformación vinculado a USAID y la guerra biológica.
La sospecha no es prueba. La prueba es una prueba y no hay prueba suficiente para ahogar. Nuestras gracias a los profesionales médicos estadounidenses que PIMPED a sí mismos al Ejército de Estados Unidos y la CIA y que nos ayudó a donde estamos ahora, una nación rota en pedazos …�VeteransToday]
Experimento de laboratorio exitoso
Entre otras cosas, el equipo de investigación estudió los murciélagos, que son las mayores incubadoras de coronavirus. Sin embargo, los murciélagos no podían transmitir el coronavirus a los humanos porque no podían interactuar con las células humanas con los receptores ACE2.
El material también indicó que los murciélagos de herradura son portadores de una cepa del coronavirus del SARS que puede transmitirse a los humanos. Se le ha denominado virus SHC014-CoV.
Para estudiar mejor este virus, los científicos copiaron el coronavirus y lo infectaron con ratones de laboratorio. Los resultados mostraron que el virus es realmente capaz de unirse a las células humanas con receptores ACE2 y multiplicarse en las células del sistema respiratorio.
En el trabajo de investigación, se observa que los materiales, muestras y equipos de laboratorio que se utilizaron en la investigación se obtuvieron del Instituto de Investigación Médica de Enfermedades Infecciosas del Ejército. Aunque todavía no es posible decir con certeza que el virus que se probó en ratones de laboratorio es el mismo que el coronavirus SARS-Cove-2.
Política de la OTAN
Sin embargo, se pueden encontrar cosas interesantes en documentos anteriores.
Por ejemplo:
- El informe de actividad de la Alianza de 2019 dice que en 2019, el primer lugar de la Alianza en investigación y desarrollo lo ocupó el tema de la protección radioquímica y biológica (29%), desplazando el problema aparentemente más urgente de Europa: el contraterrorismo (resultó ser 4 – prioridad m).
- Un año antes, en 2018, la situación era exactamente la contraria: el terrorismo, como debería ser, ocupaba el primer lugar (28%) y la protección radioquímica y biológica en el cuarto (13%).
Como escribe el soplón de Bruselas en el canal de telegramas, “dada la ausencia de razones visibles para un cambio tan brusco en los intereses científicos, hay dos opciones y ambas son desagradables:
- o la OTAN ahora menea el quinto punto, falsificando los datos para mostrar “y siempre estamos preparados para los virus, somos modernos”,
- o incluso en 2019 en la alianza, Dios me perdone, sabían de dónde vendría el problema.
Sí, la primera opción es mucho más real, pero, ya ves, los hechos son sorprendentes.
Fuente: �Pravda
Investigación original de 2015 sin editar y completa
Publicado:�09 de noviembre de 2015Un grupo similar al SARS de coronavirus de murciélagos circulantes muestra potencial para la emergencia humana por Vineet D Menachery, Boyd L Yount Jr, Kari Debbink, Sudhakar Agnihothram, Lisa E Gralinski, Jessica A Plante, Rachel L Graham, Trevor Scobey, Xing-Yi Ge, Eric F Donaldson, Scott H Randell, Antonio Lanzavecchia, Wayne A Marasco, Zhengli-Li Shi, Ralph S Baric
Nature Medicine volumen 21, páginas 1508-1513 (2015)
Se publicó una corrección de este artículo el 6 de abril de 2016
Este artículo ha sido actualizado
Resumen
La aparición del coronavirus del síndrome respiratorio agudo severo (SARS-CoV) y el síndrome respiratorio de Oriente Medio (MERS) -CoV subraya la amenaza de eventos de transmisión entre especies que conducen a brotes en humanos. Aquí examinamos el potencial de enfermedad de un virus similar al SARS, SHC014-CoV, que actualmente está circulando en las poblaciones de murciélagos de herradura chinos�1 . Usando el sistema�2 de genética inversa del SARS-CoV , generamos y caracterizamos un virus quimérico que expresa el pico del coronavirus de murciélago SHC014 en una columna vertebral del SARS-CoV adaptada al ratón.
Los resultados indican que los virus del grupo 2b que codifican el pico SHC014 en una columna vertebral de tipo salvaje pueden usar de manera eficiente múltiples ortólogos de la enzima convertidora de angiotensina humana II (ACE2) del receptor del SARS, replicarse de manera eficiente en las células primarias de las vías respiratorias humanas y lograr títulos�in vitro equivalentes a cepas epidémicas de SARS-CoV. Además, los experimentos�in vivo demuestran la replicación del virus quimérico en el pulmón del ratón con una patogénesis notable.
La evaluación de las modalidades inmunoterapéuticas y profilácticas basadas en el SRAS disponibles reveló una eficacia deficiente; Tanto el enfoque de anticuerpos monoclonales como de vacunas no lograron neutralizar ni proteger de la infección con CoV utilizando la nueva proteína de pico.
Sobre la base de estos hallazgos, volvimos a derivar sintéticamente un virus recombinante SHC014 infeccioso de longitud completa y demostramos una replicación viral robusta tanto �in vitro como �in vivo . Nuestro trabajo sugiere un riesgo potencial de reaparición del SARS-CoV de los virus que circulan actualmente en las poblaciones de murciélagos.
Principal
La aparición del SARS-CoV anunció una nueva era en la transmisión entre especies de enfermedades respiratorias graves con la globalización que condujo a una rápida propagación en todo el mundo y un impacto económico masivo�3,4 . Desde entonces, varias cepas, incluidas las cepas de influenza A H5N1, H1N1 y H7N9 y MERS-CoV, han surgido de poblaciones animales, causando enfermedades, mortalidad y dificultades económicas considerables en las regiones afectadas�5 . Aunque las medidas de salud pública pudieron detener el brote de SARS-CoV�4 , estudios recientes de metagenómica han identificado secuencias de virus similares al SARS estrechamente relacionados que circulan en las poblaciones de murciélagos chinos y que pueden representar una amenaza futura�1,6 .
Sin embargo, los datos de secuencia por sí solos proporcionan información mínima para identificar y prepararse para futuros virus prepandémicos. Por lo tanto, para examinar el potencial de emergencia (es decir, el potencial de infectar a los humanos) de los CoV de murciélago circulantes, construimos un virus quimérico que codifica una nueva proteína de pico de CoV zoonótica, a partir de la secuencia RsSHC014-CoV que se aisló de murciélagos de herradura chinos�1 —En el contexto de la columna vertebral adaptada al ratón del SARS-CoV. El virus híbrido nos permitió evaluar la capacidad de la nueva proteína de pico para causar enfermedad independientemente de otras mutaciones adaptativas necesarias en su columna vertebral natural.
Usando este enfoque, caracterizamos la infección por CoV mediada por la proteína de pico SHC014 en células primarias de las vías respiratorias humanas e �in vivo y probamos la eficacia de las terapias inmunes disponibles contra SHC014-CoV. En conjunto, la estrategia traduce datos metagenómicos para ayudar a predecir y prepararse para futuros virus emergentes.
Las secuencias de SHC014 y el RsWIV1-CoV relacionado muestran que estos CoV son los parientes más cercanos a las cepas epidémicas de SARS-CoV (Fig. 1a, b); sin embargo, existen diferencias importantes en los 14 residuos que se unen al ACE2 humano, el receptor del SARS-CoV, incluidos los cinco que son críticos para el rango de hospedadores: Y442, L472, N479, T487 e Y491 (ref. 7).
En WIV1, tres de estos residuos varían de la cepa Epidemia SARS-CoV Urbani, pero no se esperaba que alteren la unión a ACE2 (Fig. 1a, b complementaria y Tabla 1 complementaria). Este hecho se confirma tanto mediante experimentos de pseudotipificación que midieron la capacidad de los lentivirus que codifican proteínas de pico de WIV1 para entrar en células que expresan ACE2 humana (Fig. 1 complementaria) como mediante ensayos de replicación�in vitro de WIV1-CoV (ref. 1). Por el contrario, 7 de los 14 residuos de interacción ACE2 en SHC014 son diferentes de los del SARS-CoV, incluidos los cinco residuos críticos para el rango de hospedadores (Fig. Complementaria 1c y Tabla complementaria 1).
Estos cambios, junto con el fracaso de los lentivirus pseudotipados que expresan el pico SHC014 para entrar en las células (Fig. Complementaria 1d), sugirieron que el pico SHC014 no puede unirse a la ACE2 humana. Sin embargo, se han informado cambios similares en las cepas de SARS-CoV relacionadas para permitir la unión de ACE2�7,8 , lo que sugiere que se requieren pruebas funcionales adicionales para la verificación.
Por lo tanto, sintetizamos el pico de SHC014 en el contexto de la estructura principal del SARS-CoV adaptado al ratón y con capacidad de replicación (en adelante nos referiremos al CoV quimérico como SHC014-MA15) para maximizar la oportunidad de estudios de patogénesis y vacunas en ratones (Fig. Complementaria). . 2a). A pesar de las predicciones tanto del modelado basado en la estructura como de los experimentos de pseudotipado, SHC014-MA15 fue viable y se replicó a títulos altos en células Vero (Fig. 2b complementaria). Similar al SARS, SHC014-MA15 también requería una molécula de ACE2 funcional para la entrada y podía usar ortólogos ACE2 humanos, de algalia y de murciélago (Fig. Complementaria 2c, d).
Para probar la capacidad del pico de SHC014 para mediar la infección de las vías respiratorias humanas, examinamos la sensibilidad de la línea celular de las vías respiratorias epiteliales humanas Calu-3 2B4 (ref.9) a la infección y encontramos una replicación robusta de SHC014-MA15, comparable a la de SARS-CoV Urbani (Figura 1c).
Para ampliar estos hallazgos, se infectaron cultivos primarios de epitelio de las vías respiratorias humanas (HAE) y mostraron una replicación robusta de ambos virus (Fig. 1d). Juntos, los datos confirman la capacidad de los virus con el pico SHC014 para infectar las células de las vías respiratorias humanas y subrayan la amenaza potencial de la transmisión entre especies de SHC014-CoV.
Figura 1: Los virus similares al SARS se replican en las células de las vías respiratorias humanas y producen patogénesis�in vivo .
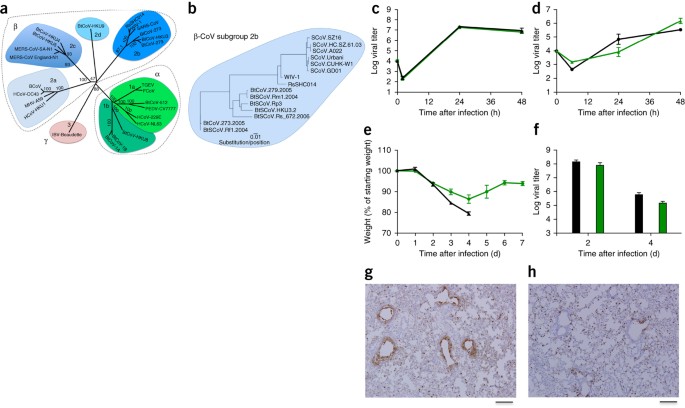
(�a ) Las secuencias del genoma de longitud completa de los CoV representativos se alinearon y mapearon filogenéticamente como se describe en los Métodos en línea. La barra de escala representa sustituciones de nucleótidos, y solo se etiqueta el soporte de arranque por encima del 70%. El árbol muestra los CoV divididos en tres grupos filogenéticos distintos, definidos como α-CoV, β-CoV y γ-CoV. Los grupos de subgrupos clásicos se marcan como 2a, 2b, 2c y 2d para los β-CoV y como 1a y 1b para los α-CoV. (�b ) Las secuencias de aminoácidos de los dominios S1 de las puntas de los β-CoV representativos del grupo 2b, incluido el SARS-CoV, se alinearon y mapearon filogenéticamente. La barra de escala representa las sustituciones de aminoácidos. (�c ,�d) Replicación viral de SARS-CoV Urbani (negro) y SHC014-MA15 (verde) después de la infección de células Calu-3 2B4 (�c ) o cultivos de células HAE de interfaz aire-líquido primarias bien diferenciadas (�d ) en una multiplicidad de infección (MOI) de 0,01 para ambos tipos de células. Las muestras se recolectaron en puntos de tiempo individuales con réplicas biológicas (�n = 3) tanto para los experimentos de Calu-3 como para los de HAE. (�e ,�f ) Pérdida de peso (�n = 9 para SARS-CoV MA15; �n = 16 para SHC014-MA15) (�e ) y replicación viral en los pulmones (�n = 3 para SARS-CoV MA15; �n = 4 para SHC014- MA15) (�f) De ratones BALB / c de 10 semanas de edad, infectados con 1 × 10�4 pfu de adaptada-ratón de SARS-CoV MA15 (negro) o SHC014-MA15 (verde) a través de la (in) ruta intranasal. (�g, h ) Se muestran imágenes representativas de secciones de pulmón teñidas para antígeno SARS-CoV N de ratones infectados con SARS-CoV MA15 (�n = 3 ratones) (�g ) o SHC014-MA15 (�n = 4 ratones) (�h ). Para cada gráfico, el valor central representa la media del grupo y las barras de error definen las barras de escala sem, 1 mm.
Imagen de tamaño completo
Para evaluar el papel del pico de SHC014 en la mediación de la infección�in vivo , infectamos ratones BALB / c de 10 semanas de edad con 10 4 unidades formadoras de placa (ufp) de SARS-MA15 o SHC014-MA15 (Fig. 1e-h). Los animales infectados con SARS-MA15 experimentaron una rápida pérdida de peso y letalidad 4 días después de la infección (dpi); por el contrario, la infección por SHC014-MA15 produjo una pérdida de peso sustancial (10%) pero no letalidad en ratones (Fig. 1e). El examen de la replicación viral reveló títulos virales casi equivalentes de los pulmones de ratones infectados con SARS-MA15 o SHC014-MA15 (Fig. 1f). Mientras que los pulmones de los ratones infectados con SARS-MA15 mostraron una tinción intensa tanto en los bronquiolos terminales como en el parénquima pulmonar 2 dpi (Fig. 1g), los de los ratones infectados con SHC014-MA15 mostraron una tinción reducida del antígeno de las vías respiratorias (Fig. 1h); por el contrario, no se observó ningún déficit en la tinción del antígeno en el parénquima o en la puntuación histológica general,
A continuación, analizamos la infección en animales de edad más susceptibles (12 meses). Los animales infectados con SARS-MA15 perdieron peso rápidamente y sucumbieron a la infección (Fig. Complementaria 3a, b). La infección por SHC014-MA15 indujo una pérdida de peso sólida y sostenida, pero tuvo una letalidad mínima. Las tendencias en la histología y los patrones de tinción de antígenos que observamos en ratones jóvenes se conservaron en los animales más viejos (Tabla complementaria 3). Excluimos la posibilidad de que SHC014-MA15 mediara la infección a través de un receptor alternativo sobre la base de experimentos con �Ace2�– / – ratones, que no mostraron pérdida de peso o tinción de antígeno después de la infección por SHC014-MA15 (Fig. 4a, b complementaria y Tabla 2 complementaria). Juntos, los datos indican que los virus con el pico SHC014 son capaces de inducir la pérdida de peso en ratones en el contexto de una columna vertebral de CoV virulenta.
Dada la eficacia preclínica de las terapias con anticuerpos monoclonales contra el ébola, como ZMApp�10 , a continuación buscamos determinar la eficacia de los anticuerpos monoclonales del SARS-CoV contra la infección por SHC014-MA15. Anteriormente se habían informado cuatro anticuerpos monoclonales humanos ampliamente neutralizantes dirigidos a la proteína de pico del SARS-CoV y son probables reactivos para la inmunoterapia�11,12,13 . Examinamos el efecto de estos anticuerpos sobre la replicación viral (expresado como porcentaje de inhibición de la replicación viral) y encontramos que mientras que el SARS-CoV Urbani de tipo salvaje fue fuertemente neutralizado por los cuatro anticuerpos a concentraciones de anticuerpos relativamente bajas (Fig. 2a-d), neutralización variada para SHC014-MA15. Fm6, un anticuerpo generado por la presentación de fagos y mutantes de escape�11,12, logró sólo niveles de fondo de inhibición de la replicación de SHC014-MA15 (Fig. 2a). De manera similar, los anticuerpos 230.15 y 227.14, que se derivaron de las células B de memoria de pacientes infectados con SARS-CoV�13, tampoco pudo bloquear la replicación de SHC014-MA15 (Fig. 2b, c). Para los tres anticuerpos, las diferencias entre las secuencias de aminoácidos de los picos de SARS y SHC014 correspondían a cambios de residuos directos o adyacentes encontrados en mutantes de escape de SARS-CoV (fm6 N479R; 230.15 L443V; 227.14 K390Q / E), lo que probablemente explica la ausencia de los anticuerpos. ‘actividad neutralizante contra SHC014. Finalmente, el anticuerpo monoclonal 109.8 pudo lograr una neutralización del 50% de SHC014-MA15, pero solo a altas concentraciones (10 μg / ml) (Fig. 2d). Juntos, los resultados demuestran que los anticuerpos ampliamente neutralizantes contra el SARS-CoV solo pueden tener una eficacia marginal contra las cepas emergentes de CoV similares al SARS, como SHC014.
Figura 2: Los anticuerpos monoclonales del SARS-CoV tienen una eficacia marginal contra los CoV similares al SARS.
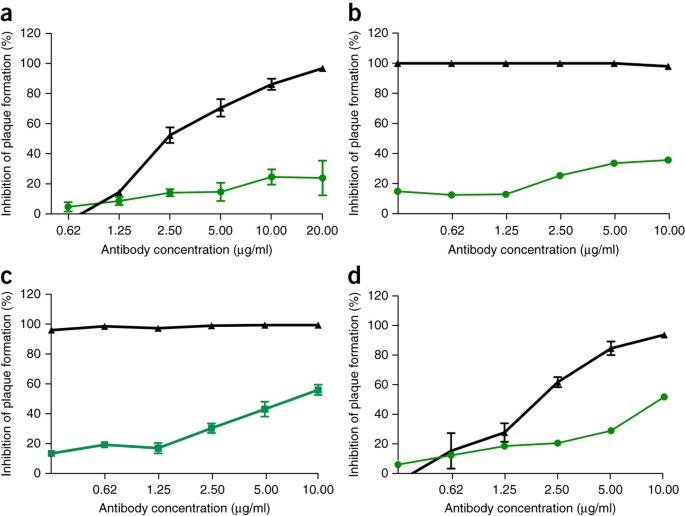
(�a –�d ) Ensayos de neutralización que evalúan la eficacia (medida como una reducción en el número de placas) de un panel de anticuerpos monoclonales, que se generaron originalmente contra SARS-CoV epidémico, contra la infección de células Vero con SARS-CoV Urbani (negro ) o SHC014-MA15 (verde). Los anticuerpos probados fueron fm6 (�n = 3 para Urbani; �n = 5 para SHC014-MA15)�11,12 (�a ), 230.15 (�n = 3 para Urbani; �n = 2 para SHC014-MA15) (�b ), 227.15 (�n = 3 para Urbani; �n = 5 para SHC014-MA15) (�c ) y 109,8 (�n = 3 para Urbani;�n = 2 para SHC014-MA15)�13 (�d ). Cada punto de datos representa la media del grupo y las barras de error definen el sem. Tenga en cuenta que las barras de error en las células Vero infectadas con SARS-CoV Urbani en �b ,�c están superpuestas por los símbolos y no son visibles.
Imagen de tamaño completo
Para evaluar la eficacia de las vacunas existentes contra la infección con SHC014-MA15, vacunamos ratones envejecidos con SARS-CoV (DIV) completo doblemente inactivado. El trabajo anterior mostró que DIV podría neutralizar y proteger a los ratones jóvenes del desafío con un virus homólogo�14 ; sin embargo, la vacuna no protegió a los animales envejecidos en los que también se observó una patología inmunitaria aumentada, lo que indica la posibilidad de que los animales resulten perjudicados por la vacunación�15 . Aquí encontramos que DIV no proporcionó protección contra el desafío con SHC014-MA15 con respecto a la pérdida de peso o el título viral (Fig. 5a, b complementaria). De acuerdo con un informe anterior con otros CoV de los grupos 2b heterólogos�15, el suero de ratones envejecidos vacunados con DIV tampoco logró neutralizar SHC014-MA15 (Fig. 5c complementaria).
En particular, la vacuna DIV resultó en una patología inmunitaria sólida (Tabla complementaria 4) y eosinofilia (Fig. Complementaria 5d-f). Juntos, estos resultados confirman que la vacuna DIV no protegería contra la infección por SHC014 y posiblemente podría aumentar la enfermedad en el grupo vacunado de edad avanzada.
En contraste con la vacunación de ratones con DIV, el uso de SHC014-MA15 como vacuna viva atenuada mostró una protección cruzada potencial contra el desafío con SARS-CoV, pero los resultados tienen importantes salvedades. Infectamos ratones jóvenes con 10�4 pfu de SHC014-MA15 y las observó durante 28 d. Luego desafiamos a los ratones con SARS-MA15 el día 29 (Fig. 6a complementaria). La infección previa de los ratones con la dosis alta de SHC014-MA15 confirió protección contra el desafío con una dosis letal de SARS-MA15, aunque solo hubo una respuesta mínima de neutralización del SARS-CoV de los antisueros provocados 28 días después de la infección por SHC014-MA15 ( Fig. Complementaria 6b, 1: 200). En ausencia de un refuerzo de antígeno secundario, 28 dpi representa el pico esperado de títulos de anticuerpos e implica que habrá una protección disminuida contra el SARS-CoV con el tiempo�16,17. Se observaron resultados similares que muestran protección frente a la exposición con una dosis letal de SARS-CoV en ratones BALB / c envejecidos con respecto a la pérdida de peso y la replicación viral (Fig. 6c, d complementaria). Sin embargo, la dosis de infección SHC014-MA15 de 10�4 pfu indujo> 10% de pérdida de peso y la letalidad en algunos animales de edad avanzada (. La Fig. 1 y complementario figura 3). Encontramos que la vacunación con una dosis más baja de SHC014-MA15 (100 ufp) no indujo pérdida de peso, pero tampoco protegió a los animales de edad avanzada de un desafío de dosis letal de SARS-MA15 (Fig. 6e, f complementaria). Juntos, los datos sugieren que el desafío SHC014-MA15 puede conferir protección cruzada contra el SARS-CoV a través de epítopos conservados, pero la dosis requerida induce patogénesis e impide su uso como vacuna atenuada.
Habiendo establecido que el pico SHC014 tiene la capacidad de mediar la infección de células humanas y causar enfermedad en ratones, a continuación sintetizamos un clon infeccioso SHC014-CoV de longitud completa basado en el enfoque utilizado para el SARS-CoV (Fig. 3a)�2 . La replicación en las células Vero no reveló ningún déficit para SHC014-CoV en relación con el de SARS-CoV (Fig. 3b); sin embargo, SHC014-CoV se atenuó significativamente (�P <0,01) en cultivos primarios de HAE tanto a las 24 como a las 48 h después de la infección (Fig. 3c).�En vivo la infección de ratones no demostró una pérdida de peso significativa, pero mostró una replicación viral reducida en los pulmones de la infección por SHC014-CoV de longitud completa, en comparación con el SARS-CoV Urbani (Fig. 3d, e). Juntos, los resultados establecen la viabilidad de SHC014-CoV de longitud completa, pero sugieren que se requiere una mayor adaptación para que su replicación sea equivalente a la del SARS-CoV epidémico en células respiratorias humanas y en ratones.
Figura 3: El SHC014-CoV de longitud completa se replica en las vías respiratorias humanas, pero carece de la virulencia del SARS-CoV epidémico.
(�a ) Esquema del clon molecular SHC014-CoV, que se sintetizó como seis ADNc contiguos (designados SHC014A, SHC014B, SHC014C, SHC014D, SHC014E y SHC014F) flanqueado por sitios BglI únicos que permitieron el ensamblaje dirigido del ADNc de longitud completa que expresa marcos de lectura abiertos (para 1a, 1b, pico, 3, envoltura, matriz, 6–8 y nucleocápside). Los nucleótidos subrayados representan las secuencias salientes formadas después de la escisión con enzimas de restricción. (�b ,�c ) Replicación viral de SARS-CoV Urbani (negro) o SHC014-CoV (verde) después de la infección de células Vero (�b ) o cultivos de células HAE de interfaz aire-líquido primarias bien diferenciadas (�c) con un MOI de 0.01. Las muestras se recolectaron en puntos de tiempo individuales con repeticiones biológicas (�n = 3) para cada grupo. Los datos representan un experimento para células Vero y HAE. (�d ,�e ) Pérdida de peso (�n = 3 para SARS-CoV MA15, �n = 7 para SHC014-CoV; �n = 6 para SARS-Urbani) (�d ) y replicación viral en los pulmones (�n = 3 para SARS-Urbani y SHC014-CoV) (�e ) de ratones de 10 semanas de edad BALB / c infectados con 1 × 10�5 pfu de SARS-CoV MA15 (gris), SHC014-CoV (verde) o SARS-CoV Urbani (negro) a través de la en ruta o en camino. Cada punto de datos representa la media del grupo y las barras de error definen el sem **P <0.01 y ***�P <0.001 usando la prueba�t de Student de dos colas de puntos de tiempo individuales.
Durante la epidemia de SARS-CoV, se establecieron rápidamente vínculos entre las civetas de palma y las cepas de CoV que se detectaron en humanos�4 .
Sobre la base de este hallazgo, el paradigma de emergencia común sostiene que el SARS-CoV epidémico se originó como un virus de murciélago, saltó a las civetas e incorporó cambios dentro del dominio de unión al receptor (RBD) para mejorar la unión a la civeta Ace2 (ref. 18).
La exposición posterior a personas en los mercados de animales vivos permitió la infección humana con la cepa de algalia, que, a su vez, se adaptó para convertirse en la cepa epidémica (Fig. 4a).
Sin embargo, el análisis filogenético sugiere que las primeras cepas humanas de SARS parecen estar más estrechamente relacionadas con las cepas de murciélago que con las cepas de civeta�18 .
Por lo tanto, un segundo paradigma sostiene que la transmisión directa murciélago-humano inició la emergencia del SARS-CoV y que las civetas de la palma sirvieron como huésped secundario y reservorio para la infección continua (Fig. 4b)�19 . Para ambos paradigmas, la adaptación de picos en un huésped secundario se considera una necesidad, y se espera que la mayoría de las mutaciones ocurran dentro del RBD, facilitando así una mejor infección. Ambas teorías implican que los grupos de CoV de murciélago son limitados y que las mutaciones en el rango de hospedadores son aleatorias y raras, lo que reduce la probabilidad de eventos de emergencia futuros en humanos.
Figura 4: Paradigmas emergentes para coronavirus.
Las cepas de coronavirus se mantienen en grupos de cuasiespecies que circulan en poblaciones de murciélagos. (�a ,�b ) Las teorías tradicionales de emergencia del SARS-CoV postulan que los mutantes del rango de hospedadores (círculo rojo) representan ocurrencias aleatorias y raras que permiten la infección de hospederos alternativos. El paradigma del huésped secundario (�a ) sostiene que un huésped no humano está infectado por un virus progenitor de murciélago y, a través de la adaptación, facilita la transmisión a los humanos; la replicación posterior en humanos conduce a la cepa viral epidémica. El paradigma directo (�b) sugiere que la transmisión se produce entre murciélagos y humanos sin el requisito de un huésped intermedio; la selección se produce entonces en la población humana con virus estrechamente relacionados que se replican en un huésped secundario, lo que permite la persistencia y adaptación viral continua en ambos. (�c ) Los datos de virus quiméricos similares al SARS sostienen que los grupos de cuasiespecies mantienen múltiples virus capaces de infectar células humanas sin necesidad de mutaciones (círculos rojos). Aunque es posible que se requieran adaptaciones en huéspedes secundarios o humanos para la aparición de una epidemia, si los virus que contienen picos SHC014 se recombinan con las cadenas principales de CoV virulentas (círculos con contornos verdes), la enfermedad epidémica puede ser el resultado en los seres humanos. Elementos de soporte de datos existentes de los tres paradigmas.
Imagen de tamaño completo
Aunque nuestro estudio no invalida las otras rutas de emergencia, sí aboga por un tercer paradigma en el que los grupos circulantes de CoV de murciélago mantienen proteínas de pico “preparadas” que son capaces de infectar a los humanos sin mutación o adaptación (Fig. 4c). Esta hipótesis se ilustra por la capacidad de un virus quimérico que contiene el pico SHC014 en una columna vertebral del SARS-CoV para causar una infección fuerte tanto en cultivos de vías respiratorias humanas como en ratones sin adaptación a RBD.
Junto con la observación de las cadenas principales de CoV patógenas previamente identificadas�3,20 , nuestros resultados sugieren que los materiales de partida necesarios para las cepas emergentes similares al SARS están circulando actualmente en reservorios animales. En particular, aunque SHC014-CoV de longitud completa probablemente requiera una adaptación de la columna vertebral adicional para mediar la enfermedad humana, los eventos de recombinación de alta frecuencia documentados en las familias de CoV subrayan la posibilidad de una emergencia futura y la necesidad de una preparación adicional.
Hasta la fecha, las pruebas genómicas de poblaciones animales se han utilizado principalmente para identificar virus nuevos en entornos de brotes�21 . El enfoque aquí amplía estos conjuntos de datos para examinar cuestiones de aparición viral y eficacia terapéutica. Consideramos que los virus con el pico SHC014 son una amenaza potencial debido a su capacidad para replicarse en cultivos primarios de vías respiratorias humanas, el mejor modelo disponible para enfermedades humanas. Además, la patogenia observada en ratones indica una capacidad de los virus que contienen SHC014 para causar enfermedades en modelos de mamíferos, sin adaptación a RBD.
En particular, el tropismo diferencial en el pulmón en comparación con el de SARS-MA15 y la atenuación de SHC014-CoV de longitud completa en cultivos de HAE en relación con el SARS-CoV Urbani sugieren que hay factores más allá de la unión de ACE2, incluida la procesividad de picos, la biodisponibilidad del receptor o el antagonismo de las respuestas inmunitarias del huésped — puede contribuir a la aparición. Sin embargo, se requieren más pruebas en primates no humanos para traducir estos hallazgos en el potencial patógeno en humanos.
Es importante destacar que el fracaso de las terapias disponibles define una necesidad crítica de estudios adicionales y de desarrollo de tratamientos. Con este conocimiento, se pueden producir programas de vigilancia, reactivos de diagnóstico y tratamientos efectivos que protejan contra la aparición de CoV específicos del grupo 2b, como SHC014, y estos se pueden aplicar a otras ramas de CoV que mantienen grupos igualmente heterogéneos.
Además de ofrecer preparación contra futuros virus emergentes, este enfoque debe considerarse en el contexto de la pausa en los estudios�de ganancia de función (GOF) impuesta por el gobierno de EE. UU . 22 .
Sobre la base de modelos de emergencia anteriores (Fig. 4a, b), no se esperaba que la creación de virus quiméricos como SHC014-MA15 aumentara la patogenicidad. Aunque SHC014-MA15 está atenuado en relación con su SARS-CoV adaptado al ratón parental, estudios similares que examinaron la patogenicidad de los CoV con el pico Urbani de tipo salvaje dentro de la columna vertebral MA15 no mostraron pérdida de peso en ratones y una replicación viral reducida�23 . Por lo tanto, en relación con el pico de Urbani – MA15 CoV, SHC014-MA15 se muestra nuevamente en la patogénesis (Fig. 1).
Sobre la base de estos hallazgos, los paneles de revisión científica pueden considerar que estudios similares que construyen virus quiméricos basados en cepas circulantes son demasiado riesgosos de realizar, ya que no se puede excluir una mayor patogenicidad en modelos de mamíferos.
Junto con las restricciones sobre cepas adaptadas a ratones y el desarrollo de anticuerpos monoclonales utilizando mutantes de escape, la investigación sobre la aparición de CoV y la eficacia terapéutica puede verse seriamente limitada en el futuro. Juntos, estos datos y restricciones representan una encrucijada de preocupaciones de investigación del GOF; El potencial para prepararse y mitigar futuros brotes debe sopesarse con el riesgo de crear patógenos más peligrosos. Al desarrollar políticas en el futuro, es importante considerar el valor de los datos generados por estos estudios y si estos tipos de estudios de virus quiméricos justifican una mayor investigación frente a los riesgos inherentes involucrados.
En general, nuestro enfoque ha utilizado datos de metagenómica para identificar una amenaza potencial planteada por el CoV SHC014 similar al SARS de murciélago circulante. Debido a la capacidad de los virus quiméricos SHC014 para replicarse en cultivos de vías respiratorias humanas, causar patogénesis �in vivo y escapar de las terapias actuales, existe una necesidad tanto de vigilancia como de terapias mejoradas contra virus circulantes similares al SARS. Nuestro enfoque también desbloquea el uso de datos metagenómicos para predecir la aparición de virus y aplicar este conocimiento en la preparación para tratar futuras infecciones por virus emergentes.
Métodos
Virus, células, infección�in vitro y ensayos de placa
Se cultivaron SARS-CoV de tipo salvaje (Urbani), SARS-CoV adaptado al ratón (MA15) y CoV quiméricos similares al SARS en células Vero E6 (obtenidas del Instituto de Investigación Médica de Enfermedades Infecciosas del Ejército de los Estados Unidos), cultivadas en Eagle modificado de Dulbecco. medio (DMEM) (Gibco, CA) y suero de clon fetal (FCS) al 5% (Hyclone, South Logan, UT) junto con antibiótico / antimicótico (Gibco, Carlsbad, CA). Las células DBT (laboratorio de Baric, fuente desconocida) que expresan ortólogos de�ACE2 se han descrito previamente tanto para humanos como para civetas;�La secuencia de bat Ace2 se basó en la de �Rhinolophus leschenaulti , y las células DBT que expresan bat �Ace2 se establecieron como se describió anteriormente�8 .
Los experimentos de pseudotipado fueron similares a los que utilizaron un pseudovirus basado en el VIH, preparado como se describió previamente�10 y examinado en células HeLa (Instituto de Virología de Wuhan) que expresaban ortólogos�ACE2 . Las células HeLa se cultivaron en medio esencial mínimo (MEM) (Gibco, CA) suplementado con FCS al 10% (Gibco, CA) como se describió previamente [�24] .
Las curvas de crecimiento en Vero E6, DBT, Calu-3 2B4 y células epiteliales primarias de las vías respiratorias humanas se realizaron como se describió anteriormente�8,25 . Ninguna de las cepas de líneas celulares de trabajo fue autenticada o probada para micoplasma recientemente, aunque las cepas de semillas originales utilizadas para crear las cepas de trabajo están libres de contaminación. Los pulmones humanos para cultivos de AEH se obtuvieron bajo los protocolos aprobados por la Junta de Revisión Institucional de la Universidad de Carolina del Norte en Chapel Hill. Los cultivos de HAE representan el epitelio de las vías respiratorias humanas altamente diferenciado que contiene células epiteliales ciliadas y no ciliadas, así como células caliciformes. Los cultivos también se cultivan en una interfaz aire-líquido durante varias semanas antes de su uso, como se describió anteriormente�26 .
Brevemente, las células se lavaron con PBS y se inocularon con el virus o se diluyeron de forma simulada en PBS durante 40 min a 37ºC. Después de la inoculación, las células se lavaron tres veces y se añadió un medio nuevo para indicar el tiempo “0”. Se recolectaron tres o más réplicas biológicas en cada punto de tiempo descrito. No se utilizó cegamiento en ninguna colección de muestras ni se asignaron muestras al azar. Todo el cultivo de virus se realizó en un laboratorio de nivel de bioseguridad (BSL) 3 con ventiladores redundantes en los gabinetes de bioseguridad, como se describió previamente por nuestro grupo�2 . Todo el personal usaba respiradores purificadores de aire motorizados (Breathe Easy, 3M) con trajes, delantales y botines Tyvek y usaba dos guantes.
Agrupación de secuencias y modelado estructural.
Las secuencias genómicas de longitud completa y las secuencias de aminoácidos de los dominios S1 del pico de CoV representativos se descargaron de Genbank o Pathosystems Resource Integration Center (PATRIC), se alinearon con ClustalX y se compararon filogenéticamente utilizando una estimación de máxima verosimilitud usando 100 bootstraps o por utilizando�el paquete PhyML , respectivamente. El árbol se generó utilizando la máxima probabilidad con el paquete PhyML. La barra de escala representa sustituciones de nucleótidos. Solo se etiquetan los nodos con soporte de arranque superior al 70%.
El árbol muestra que los CoV se dividen en tres grupos filogenéticos distintos definidos como α-CoV, β-CoV y γ-CoV. Los clústeres de subgrupos clásicos se marcan como 2a, 2b, 2c y 2d para los β-CoV, y 1a y 1b para los α-CoV. Los modelos estructurales se generaron utilizando Modeller (Max Planck Institute Bioinformatics Toolkit) para generar modelos de homología para SHC014 y Rs3367 del SARS RBD en complejo con ACE2 basado en la estructura cristalina 2AJF (Protein Data Bank). Los modelos de homología se visualizaron y manipularon en MacPyMol (versión 1.3).
Construcción de virus quiméricos similares al SARS.
Tanto los virus de tipo salvaje como los quiméricos se derivaron de SARS-CoV Urbani o del correspondiente clon infeccioso adaptado al ratón (SARS-CoV MA15) (ic) como se describió previamente�27 .
Los plásmidos que contenían secuencias de picos para SHC014 se extrajeron mediante digestión de restricción y se ligaron en el plásmido E y F del clon infeccioso MA15. El clon se diseñó y adquirió de Bio Basic como seis ADNc contiguos usando secuencias publicadas flanqueadas por sitios únicos de endonucleasa de restricción de clase II (BglI). Posteriormente, los plásmidos que contienen fragmentos de genoma de SARS-CoV quimérico y SHC014-CoV de tipo salvaje se amplificaron, escindieron, ligaron y purificaron.
Luego, se realizaron reacciones de transcripción in vitro para sintetizar ARN genómico de longitud completa, que se transfectó en células Vero E6 como se describió anteriormente�2 . El medio de las células transfectadas se recogió y sirvió como reservas de semillas para experimentos posteriores. Los virus quiméricos y de longitud completa se confirmaron mediante análisis de secuencia antes de su uso en estos estudios. La construcción sintética del mutante quimérico y SHC014-CoV de longitud completa fue aprobada por el Comité de Bioseguridad Institucional de la Universidad de Carolina del Norte y el Comité de Investigación de Preocupación de Uso Dual.
Declaración de Ética
Este estudio se llevó a cabo de acuerdo con las recomendaciones para el cuidado y uso de animales de la Oficina de Bienestar Animal de Laboratorio (OLAW), NIH. El Comité Institucional de Uso y Cuidado de Animales (IACUC) de la Universidad de Carolina del Norte en Chapel Hill (UNC, Número de permiso A-3410-01) aprobó el protocolo de estudio animal (IACUC # 13-033) utilizado en estos estudios.
Ratones e infección�in vivo
Se encargaron ratones BALB / cAnNHsD hembra, de 10 semanas y de 12 meses de edad de Harlan Laboratories. Las infecciones de ratón se realizaron como se describió anteriormente [�20] . Brevemente, los animales se llevaron a un laboratorio BSL3 y se dejaron aclimatar durante 1 semana antes de la infección. Para la infección y la vacunación con virus vivo atenuado, los ratones se anestesiaron con una mezcla de ketamina y xilazina y se infectaron por vía intranasal, cuando se expusieron, con 50 μl de solución salina tamponada con fosfato (PBS) o virus diluido con tres o cuatro ratones por punto de tiempo, por grupo de infección por dosis como se describe en las leyendas de las figuras.
En el caso de ratones individuales, las anotaciones de infección, incluida la falta de inhalación de la dosis completa, el burbujeo del inóculo por la nariz o la infección a través de la boca, pueden haber llevado a la exclusión de los datos del ratón a discreción del investigador; posinfección, no se definen otros criterios de exclusión o inclusión preestablecidos. No se utilizó cegamiento en ningún experimento con animales y los animales no se asignaron al azar. Para la vacunación, se vacunaron ratones jóvenes y ancianos mediante inyección en la planta de la pata con un volumen de 20 µl de 0,2 µg de vacuna de SARS-CoV con doble inactivación con alumbre o PBS simulado; A continuación, los ratones se reforzaron con el mismo régimen 22 días después y se desafiaron 21 días después.
Para todos los grupos, según el protocolo, los animales fueron monitoreados diariamente para detectar signos clínicos de enfermedad (encorvamiento, pelaje erizado y actividad reducida) durante la duración del experimento. La pérdida de peso se controló diariamente durante los primeros 7 días, después de lo cual el control del peso continuó hasta que los animales recuperaron su peso inicial inicial o mostraron un aumento de peso de forma continua durante 3 días.
Todos los ratones que perdieron más del 20% de su peso corporal inicial fueron alimentados en tierra y monitoreados varias veces al día, siempre que estuvieran por debajo del límite del 20%. Los ratones que perdieron más del 30% de su peso corporal inicial se sacrificaron inmediatamente según el protocolo. Cualquier ratón que se considerara moribundo o con poca probabilidad de recuperarse también fue sacrificado humanamente a discreción del investigador. La eutanasia se realizó mediante una sobredosis de isoflurano y la muerte se confirmó por dislocación cervical. Todos los estudios con ratones se realizaron en la Universidad de Carolina del Norte (Garantía de Bienestar Animal # A3410-01) utilizando protocolos aprobados por el Comité Institucional de Uso y Cuidado de Animales de la UNC (IACUC).
Análisis histológico.
Se extrajo el pulmón izquierdo y se sumergió en formalina tamponada al 10% (Fisher) sin inflar durante 1 semana. Los tejidos se incrustaron en parafina y las instalaciones centrales de histopatología del UNC Lineberger Comprehensive Cancer Center prepararon secciones de 5 μm. Para determinar la extensión de la tinción del antígeno, las secciones se tiñeron para el antígeno viral usando un anticuerpo anti-nucleocápsida del SARS-CoV policlonal disponible comercialmente (Imgenex) y se puntuaron de manera ciega para la tinción de las vías respiratorias y el parénquima como se describió anteriormente�20 . Las imágenes se capturaron utilizando un microscopio Olympus BX41 con una cámara Olympus DP71.
Ensayos de neutralización de virus.
Los ensayos de títulos de neutralización por reducción de placa se realizaron con anticuerpos previamente caracterizados contra el SARS-CoV, como se describió anteriormente. 11,
12,13
. Brevemente, los anticuerpos neutralizantes o el suero se diluyeron en serie dos veces y se incubaron con 100 ufp de los diferentes clones infecciosos, cepas de SARS-CoV durante 1 ha 37ºC. Los virus y los anticuerpos se añadieron a continuación a una placa de 6 pocillos con 5 x 10�5 células Vero E6 / pocillo con múltiples repeticiones ( n ≥ 2). Después de una incubación de 1 hora a 37 ° C, las células se cubrieron con 3 ml de agarosa al 0,8% en un medio. Las placas se incubaron durante 2 días a 37 ° C, se tiñeron con rojo neutro durante 3 horas y se contaron las placas. El porcentaje de reducción de placa se calculó como (1 – (número de placas con un anticuerpo / número de placas sin anticuerpo)) x 100.
análisis estadístico
Todos los experimentos se realizaron contrastando dos grupos experimentales (ya sea dos virus o cohortes vacunados y no vacunados). Por lo tanto, las diferencias significativas en el título viral y la puntuación histológica se determinaron mediante una t de Student de dos colas� prueba Student de en puntos de tiempo individuales. Los datos se distribuyeron normalmente en cada grupo que se estaba comparando y tuvieron una varianza similar.
Bioseguridad y bioseguridad
Los estudios informados se iniciaron después de que el Comité Institucional de Bioseguridad de la Universidad de Carolina del Norte aprobó el protocolo experimental (Título del proyecto: Generación de clones infecciosos de CoV similares al SARS de murciélago; ID del plan de seguridad del laboratorio: 20145741; ID del anexo G: 12279).
Estos estudios se iniciaron antes de la pausa de financiación de la investigación del proceso deliberativo del gobierno de los�EE. UU. Sobre investigaciones seleccionadas de ganancia de función que involucran virus de influenza, MERS y SARS . Este documento ha sido revisado por la agencia financiadora, NIH. Se solicitó la continuación de estos estudios, y esto ha sido aprobado por los NIH.
SARS-CoV es un agente selecto. Todo el trabajo para estos estudios se realizó con procedimientos operativos estándar (SOP) aprobados y condiciones de seguridad para SARS-CoV, MERs-CoV y otros CoV relacionados. Nuestras instalaciones institucionales CoV BSL3 han sido diseñadas para cumplir con los requisitos de seguridad que se recomiendan en Bioseguridad en Laboratorios Microbiológicos y Biomédicos (BMBL), el Departamento de Salud y Servicios Humanos de EE. UU., El Servicio de Salud Pública, los Centros para el Control de Enfermedades (CDC). ) y los NIH. Los planes de seguridad del laboratorio se enviaron al Departamento de Salud y Seguridad Ambiental (EHS) y los CDC, y la instalación ha sido aprobada para su uso. Se requiere acceso con tarjeta electrónica para ingresar a la instalación.
Todos los trabajadores han sido capacitados por EHS para usar de manera segura respiradores purificadores de aire (PAPR), y se han implementado hábitos de trabajo apropiados en una instalación BSL3 y planes de vigilancia médica activa. Nuestras instalaciones CoV BSL3 contienen ventiladores redundantes, energía de emergencia para ventiladores y gabinetes y congeladores de seguridad biológica, y nuestras instalaciones pueden acomodar bastidores para mouse SealSafe. Los materiales clasificados como agentes BSL3 consisten en SARS-CoV, cepas precursoras de murciélago CoV, MERS-CoV y mutantes derivados de estos patógenos. Dentro de las instalaciones de BSL3, la experimentación con un virus infeccioso se realiza en una cabina de bioseguridad (BSC) de Clase II certificada.
Todos los miembros del personal usan uniformes médicos, trajes y delantales Tyvek, PAPR y cubrezapatos, y sus manos tienen dos guantes. Los usuarios de BSL3 están sujetos a un plan de vigilancia médica supervisado por la Clínica de Salud Ocupacional para Empleados Universitarios (UEOHC), que incluye un examen físico anual, la vacunación anual contra la influenza y el informe obligatorio de cualquier síntoma asociado con la infección por CoV durante los períodos de trabajo en el BSL3. Todos los usuarios de BSL3 están capacitados en el manejo de la exposición y los protocolos de notificación, están preparados para la auto cuarentena y han sido capacitados para la entrega segura a un departamento local de manejo de enfermedades infecciosas en una situación de emergencia. Todos los eventos de exposición potencial son informados e investigados por EHS y UEOHC, con informes presentados tanto a los CDC como a los NIH.
Códigos de adhesión
Adhesiones
Banco de datos de proteínas
- 2AJF
Cambia la historia
20 de noviembre de 2015
En la versión de este artículo publicada inicialmente en línea, los autores omitieron reconocer una fuente de financiamiento, financiamiento de USAID-EPT-PREDICT de EcoHealth Alliance, a Z.-LS El error se ha corregido para las versiones impresas, PDF y HTML de este artículo. .
Referencias
- Ge, XY y col. Aislamiento y caracterización de un coronavirus similar al SARS de murciélago que utiliza el receptor ACE2.�Nature� 503 , 535–538 (2013).
- CAS
- Artículo
- Google Académico
- Yount, B. y col. Genética inversa con un ADNc infeccioso de longitud completa del coronavirus del síndrome respiratorio agudo severo.�Proc. Natl. Acad. Sci. USA� 100 , 12995-13000 (2003).
-
Becker, MM y col. El coronavirus sintético recombinante de murciélago similar al SARS es infeccioso en células cultivadas y en ratones.�Proc. Natl. Acad. Sci. USA� 105 , 19944–19949 (2008).
- Peiris, JS, Guan, Y. & Yuen, KY Síndrome respiratorio agudo severo.�Nat. Medicina.� 10 , S88-S97 (2004).
- CAS
- Artículo
- Google Académico
- Al-Tawfiq, JA y col. Vigilancia de virus respiratorios emergentes.�Lancet Infect. Dis.� 14 , 992–1000 (2014).
- Él, B. et al. Identificación de diversos alfacoronavirus y caracterización genómica de un nuevo coronavirus similar al síndrome respiratorio agudo severo de murciélagos en China.�J. Virol.� 88 , 7070–7082 (2014).
- Artículo
- Google Académico
- Li, F.Reconocimiento de receptores e infecciones entre especies del coronavirus del SARS.�Antiviral Res.� 100 , 246-254 (2013).
- CAS
- Artículo
- Google Académico
- Sheahan, T. et al. Mecanismos de expansión del rango de hospedadores del coronavirus del síndrome respiratorio agudo severo zoonótico en el epitelio de las vías respiratorias humanas.�J. Virol.� 82 , 2274–2285 (2008).
- CAS
- Artículo
- Google Académico
- Yoshikawa, T. et al. Respuestas inmunitarias innatas dinámicas de las células epiteliales bronquiales humanas a la infección por coronavirus asociada al síndrome respiratorio agudo grave.�PLoS ONE� 5 , e8729 (2010).
- Qiu, X. y col. Reversión de la enfermedad avanzada por el virus del Ébola en primates no humanos con ZMapp.�Nature� 514 , 47–53 (2014).
- CAS
- Artículo
- Google Académico
- Sui, J. y col. Ampliación de la actividad de neutralización para bloquear directamente una vía de evolución dominante del coronavirus del SARS impulsada por anticuerpos.�PLoS Pathog.� 4 , e1000197 (2008).
- Sui, J. y col. Efectos de los anticuerpos del dominio de unión al receptor de proteína anti-espiga humana sobre el escape y la aptitud para la neutralización del coronavirus del síndrome respiratorio agudo severo.�J. Virol.� 88 , 13769-13780 (2014).
- Artículo
- Google Académico
- Rockx, B. y col. Escape de los efectos de neutralización de anticuerpos monoclonales humanos�in vitro e �in vivo aptitud del coronavirus del síndrome respiratorio agudo severo.�J. Infect. Dis.� 201 , 946–955 (2010).
- Spruth, M. y col. Una vacuna de coronavirus del SARS candidato a virus completo doblemente inactivado estimula las respuestas de anticuerpos neutralizantes y protectores.�Vaccine� 24 , 652–661 (2006).
- Bolles, M. y col. Una vacuna contra el coronavirus del síndrome respiratorio agudo severo doblemente inactivada proporciona una protección incompleta en ratones e induce una mayor respuesta pulmonar proinflamatoria eosinofílica tras la exposición.�J. Virol.� 85 , 12201–12215 (2011).
- CAS
- Artículo
- Google Académico
- Siegrist, C.-A. en�Vacunas 6ª ed. (eds. Plotkin, SA, Orenstein, WA y Offit, PA) 14–32 (WB Saunders, 2013).
- Deming, D. y col. Eficacia de la vacuna en ratones senescentes desafiados con SARS-CoV recombinante que porta variantes de picos epidémicos y zoonóticos.�PLoS Med.� 3 , e525 (2006).
- Graham, RL, Donaldson, EF y Baric, RS Una década después del SARS: estrategias para controlar los coronavirus emergentes.�Nat. Rev. Microbiol.� 11 , 836–848 (2013).
- CAS
- Artículo
- Google Académico
- Graham, RL & Baric, RS Recombination, reservorios y el pico modular: mecanismos de transmisión cruzada de coronavirus.�J. Virol.� 84 , 3134–3146 (2010).
- Agnihothram, S. et al. Un modelo de ratón para el subgrupo 2c de betacoronavirus usando una variante de la cepa HKU5 de coronavirus de murciélago.�MBio� 5 , e00047-14 (2014).
- Relman, DA Metagenómica, diagnóstico de enfermedades infecciosas e investigaciones de brotes: ¿secuencia primero, pregunta después?�Mermelada. Medicina. Assoc.� 309 , 1531-1532 (2013).
- Kaiser, J. La moratoria sobre estudios de virología de riesgo deja el trabajo en 14 instituciones en el limbo.�ScienceInsider� http://news.sciencemag.org/biology/2014/11/moratorium-risky-virology-studies-leaves-work-14-institutions-limbo (2014).
-
Frieman, M. y col. Determinantes moleculares de la patogénesis y virulencia del coronavirus del síndrome respiratorio agudo severo en modelos de enfermedad humana en ratones jóvenes y envejecidos.�J. Virol.� 86 , 884–897 (2012).
-
Ren, W. y col. Diferencia en el uso de receptores entre el coronavirus del síndrome respiratorio agudo severo (SARS) y el coronavirus similar al SARS de origen murciélago.�J. Virol.� 82 , 1899-1907 (2008).
- CAS
- Artículo
- Google Académico
-
Sims, AC y col. La liberación del bloqueo de importación nuclear del coronavirus del síndrome respiratorio agudo severo mejora la transcripción del huésped en las células pulmonares humanas.�J. Virol.� 87 , 3885–3902 (2013).
-
Fulcher, ML, Gabriel, S., Burns, KA, Yankaskas, JR & Randell, SH Cultivos de células epiteliales de las vías respiratorias humanas bien diferenciadas.�Methods Mol. Medicina.� 107 , 183–206 (2005).
- Roberts, A. y col. Un coronavirus del SARS adaptado al ratón causa enfermedad y mortalidad en ratones BALB / c.�PLoS Pathog.� 3 , e5.
Expresiones de gratitud
La investigación en este manuscrito fue apoyada por subvenciones del Instituto Nacional de Alergias y Enfermedades Infecciosas y el Instituto Nacional de Envejecimiento de los Institutos Nacionales de Salud de EE. UU. (NIH) bajo los premios U19AI109761 (RSB), U19AI107810 (RSB), AI085524 (WAM), F32AI102561 (VDM) y K99AG049092 (VDM), y por los premios de la Fundación Nacional de Ciencias Naturales de China 81290341 (Z.-LS) y 31470260 (X.-YG), y por fondos de USAID-EPT-PREDICT de EcoHealth Alliance (Z. -LS). Los cultivos epiteliales de las vías respiratorias humanas fueron apoyados por el Instituto Nacional de Diabetes y Enfermedades Digestivas y Renales de los NIH bajo el premio NIH DK065988 (SHR). También agradecemos a MT Ferris (Departamento de Genética, Universidad de Carolina del Norte) por la revisión de los enfoques estadísticos y CT Tseng (Departamento de Microbiología e Inmunología, University of Texas Medical Branch) por proporcionar células Calu-3. Los experimentos con los virus recombinantes SHC014 quiméricos y de longitud completa se iniciaron y realizaron antes de que se detuviera la financiación de la investigación del GOF y desde entonces han sido revisados y aprobados para su estudio continuo por parte de los NIH. El contenido es responsabilidad exclusiva de los autores y no necesariamente representa las opiniones oficiales de los NIH.
Información del autor
Afiliaciones
- Departamento de Microbiología e Inmunología, Universidad de Carolina del Norte en Chapel Hill, Chapel Hill, Carolina del Norte, EE. UU.
- Kari Debbink
- Y Ralph S Baric
- Laboratorio clave de patógenos especiales y bioseguridad, Instituto de Virología de Wuhan, Academia de Ciencias de China, Wuhan, China
- Xing-Yi Ge
- Y Zhengli-Li Shi
- Departamento de Biología y Fisiología Celular, Universidad de Carolina del Norte en Chapel Hill, Chapel Hill, Carolina del Norte, EE. UU.
- Scott H. Randell
- Centro de Fibrosis Quística, Marsico Lung Institute, Universidad de Carolina del Norte en Chapel Hill, Chapel Hill, Carolina del Norte, EE. UU.
- Scott H. Randell
- Instituto de Investigación en Biomedicina, Instituto Bellinzona de Microbiología, Zúrich, Suiza
- Antonio Lanzavecchia
- Departamento de Inmunología del Cáncer y SIDA, Dana-Farber Cancer Institute, Harvard Medical School, Boston, Massachusetts, EE. UU.
- Wayne A Marasco
- Departamento de Medicina, Facultad de Medicina de Harvard, Boston, Massachusetts, EE. UU.
- Wayne A Marasco
Contribuciones
VDM diseñó, coordinó y realizó experimentos, completó el análisis y redactó el manuscrito. BLY diseñó el clon infeccioso y recuperó virus quiméricos; SA completó los ensayos de neutralización; LEG ayudó a realizar experimentos con ratones; TS y JAP completaron experimentos con ratones y ensayos de placa; X.-YG realizó experimentos de pseudotipado; KD generó predicciones y figuras estructurales; Análisis filogenético generado por EFD; RLG completó el análisis de ARN; SHR proporcionó cultivos primarios de HAE; AL y WAM proporcionaron reactivos de anticuerpos monoclonales críticos; y Z.-LS proporcionó plásmidos y secuencias de espigas SHC014. RSB diseñó experimentos y escribió el manuscrito.
Autores correspondientes
Correspondencia a Vineet D Menachery o Ralph S Baric.
Declaraciones de ética
Conflicto de intereses
Los autores declaran no tener intereses económicos en competencia.
Texto y figuras complementarias
Figuras complementarias 1 a 6 y tablas complementarias 1 a 4 (PDF 4747 kb)
Reimpresiones y permisos
Citar este artículo
Menachery, V., Yount, B., Debbink, K. �et al.� Un grupo similar al SARS de coronavirus de murciélagos circulantes muestra potencial para la emergencia humana.�Nat Med� 21,� 1508-1513 (2015). https://doi.org/10.1038/nm.3985
- Recibió12 de junio de 2015
- Aceptado08 de octubre de 2015
- Publicado09 de noviembre de 2015
- Fecha de asuntoDiciembre de 2015
Fuentes:
- Un grupo de coronavirus de murciélago similar al SARS muestra potencial para la emergencia humana
- Aislamiento y caracterización de un coronavirus de murciélago similar al SARS que utiliza el receptor ACE2
- Síndrome respiratorio agudo severo
- Reversión de la enfermedad avanzada por el virus del Ébola en primates no humanos con ZMapp
- Una década después del SARS: estrategias para controlar los coronavirus emergente
